转录因子和组蛋白通过与DNA相互作用(DNA-Protein Interaction,DPI)发挥着调控基因复制、表达、重组和修复的重要作用,在实现细胞功能的过程中至关重要。其中转录因子组合决定了细胞的增值、分化以及死亡,是表观基因组学研究的重要组成部分。
研究DNA-蛋白质相互作用的实验方法主要有凝胶阻滞实验、DNase 1 足迹实验、甲基化干扰实验、体内足迹实验和ChIP-seq等。基于NGS研究手段的ChIP-seq技术可以真实反应靶蛋白在全基因组上的结合情况,是非常经典的DNA结合位点分析方法,至今仍然在广泛使用。
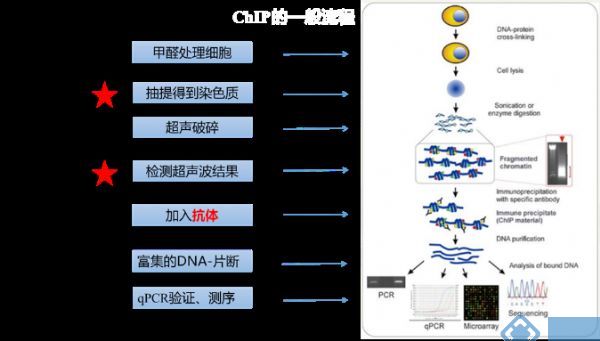
ChIP-Seq的基本实验流程过程包括(图1):甲醛交联将DNA结合蛋白和DNA紧密固定;然后抽提染色质,超声破碎DNA断裂,片段范围在200-500bp之间;抗体孵育结合靶转录因子,Protein A/G磁珠拉取抗体-蛋白-DNA复合物;蛋白消化后再通过PCR、芯片或者二代测序对DNA序列进行鉴定。
图1 ChIP-seq原理示意图[1]
看似简单,实则有几个技术难题,如果实验条件摸索不成熟会消耗大量时间。
首先,ChIP需要较多的细胞量,免疫共沉淀需要足够的靶标蛋白,而靶标蛋白有限的情况下就需要提供大量的细胞;
其次,信噪比低,很多转录因子和染色体的结合相对松散,需要甲醛强交联,以防止后续洗涤过程破坏这些结合,这样导致很多非特异性信号,需要更高的数据量得到真实peak值;
此外,实验的打断条件、重复性差等都需要系统的摸索才能得到较好的结果。
这些因素综合起来导致ChIP对新手非常不友好,需要采购特定的设备、很长时间的预实验才能得到满意的结果。
为了克服这些难题,CUT&Run以及更加成熟CUT&Tag被开发出来。2019年,美国弗雷德哈钦森癌症研究中心的 Henikoff 博士在 Nature Communication 公开了 CUT&Tag 技术的详细结果与实验方案。
CUT&Tag(Cleavage Under Targets and Tagmentation, CUT&Tag)技术,它免去了甲醛交联、超声破碎和免疫共沉淀的过程,这样既节省了初始实验材料又提高了信噪比、提升了实验重复性。
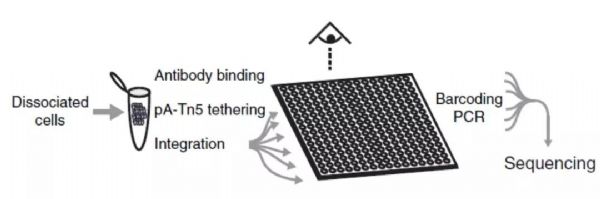
基本技术流程包括(图2):首先,特异性抗体和靶标蛋白孵育结合;加入Tn5转座酶—Protein A复合物,其中Tn5转座酶两端已装载好建库接头引物;特异性抗体和Protein A相结合,转座酶Tn5被“拉”到转录因子附近;转座酶Tn5一次性完成附近DNA打断并完成NGS加接头过程,可直接建库[2]。基于CUT&Tag,单细胞转录因子结合位点的研究也成为可能[2],这也展示了CUT-Tag的潜能。
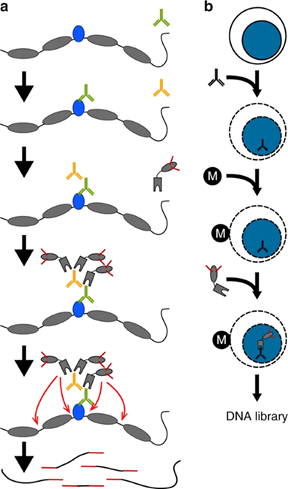
图2 CUT&Tag原理示意图[1]
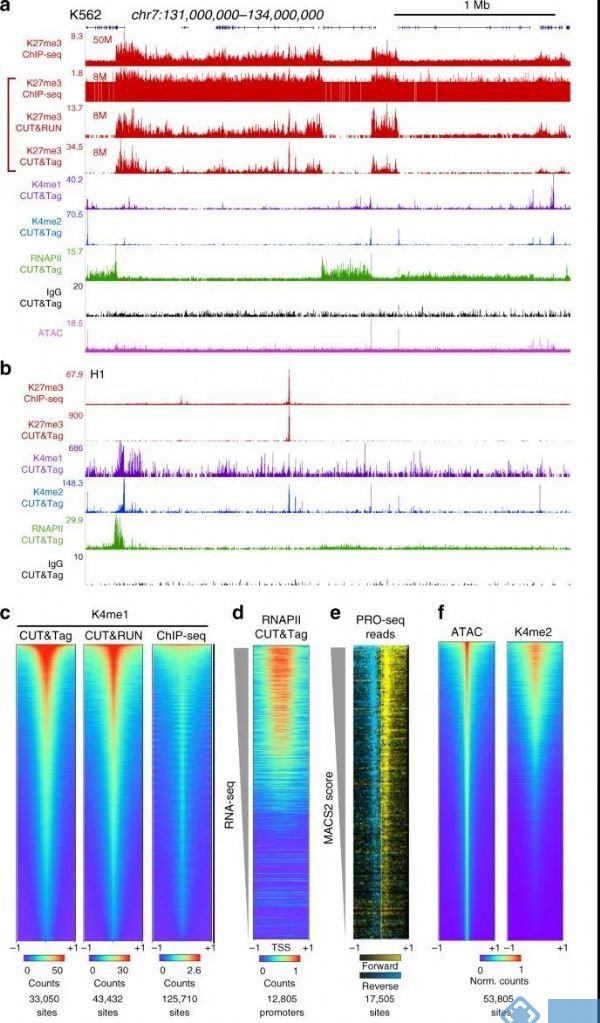
为证明Cut&Tag技术的有效性,Henikoff 博士在对组蛋白H3K27me3进行研究时对比使用了ChIP-seq、Cut&RUN、Cut&Tag三种方法。从不同角度对三组数据进行比较后,发现Cut&Tag技术具有显著优势,具体结果如下:
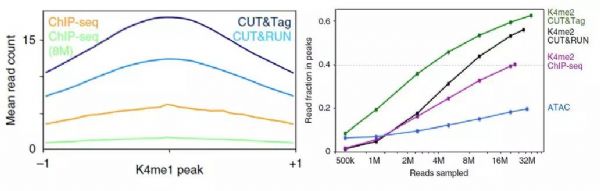
1. CUT&Tag 具有更好的信噪比和更低的背景
图3 ChIP-Seq 、CUT&RUN、CUT&Tag实验结果对比
2. CUT&Tag 在识别染色质特征时最高效
3. 重复性好,灵敏度高
图5 a:CUT&Tag、CUT&RUN、ChIP-Seq三种方法分析H3K4me1组蛋白修饰:CUT&Tag在识别染色质特征时最有效,能够给出ChIP-Seq无法读出的信息;b:peak-Calling的对比,使用默认参数调用每种方法上的峰值,结果显示CUT&Tag比CUT&RUN、ChIP-seq或ATAC-seq有更高的信号。
4. CUT&Tag 能对极少量细胞甚至单细胞进行分析
CUT&Tag 能够对极少量细胞(60 个细胞)甚至单细胞进行分析。由于 PCR 之前的反应都在细胞内进行,Henikoff 博士通过分配单个细胞到 5184 纳米孔,再加入带随机标签的引物进行扩增的办法,实现单细胞测序。
图6 CUT&Tag 单细胞测序流程
一个好的抗体是CUT&Tag成功的前提!这个抗体要能识别天然构象蛋白且特异性强,最好选用文献验证过的抗体,实验前并进行IP验证。如果实在没有合适的抗体,可以选择标签蛋白,如FLAG和HA等经过有效验证的单克隆抗体。